
中国水稻科学 ›› 2015, Vol. 29 ›› Issue (3): 327-334.DOI: 10.3969/j.issn.1001G7216.2015.03.013
• 研究报告 • 上一篇
王玲1,2, 左示敏1, 张亚芳1, 陈宗祥1, 潘学彪1,*( ), 黄世文2,*(
), 黄世文2,*( )
)
收稿日期:2014-07-31
修回日期:2014-10-16
出版日期:2015-05-10
发布日期:2015-05-10
通讯作者:
潘学彪,黄世文
基金资助:
Ling WANG1,2, Shi-min ZUO1, Ya-fang ZHANG1, Zong-xiang CHEN1, Xue-biao PAN1,*( ), Shi-wen HUANG2,*(
), Shi-wen HUANG2,*( )
)
Received:2014-07-31
Revised:2014-10-16
Online:2015-05-10
Published:2015-05-10
Contact:
Xue-biao PAN, Shi-wen HUANG
摘要:
由稻瘟病菌(Magnaporthe oryzae)侵染引起的稻瘟病是全球水稻生产上最严重的病害之一。利用6对SSR荧光标记对采自四川绵阳、营山、雅安、北川和武胜地区的5个稻瘟病菌群体的遗传结构进行分析。结果表明,在124个稻瘟病菌中检测出43个等位基因,平均每个位点的观测等位基因数为7.2,有效等位基因数为3.1,所有位点均显著偏离Hardy-Weinberg平衡。5个群体的平均观测杂合度(0.374)低于期望杂合度(0.502),暗示群体内存在因近交而导致的杂合子缺失。AMOVA分析显示,绝大多数遗传变异(81.17%)存在于群体内个体间,仅有18.83%的变异来自于群体间的差异。5个地理群体间呈现高水平的遗传分化(遗传分化系数为0.057~0.528)。Mantel检验表明,群体间的遗传距离与地理距离相关未达显著水平,说明稻瘟病菌的遗传变异呈现随机分布的空间模式。群体遗传学数据分析表明5个群体间存在不同程度的基因流(基因流水平为0.472~4.347),基于贝叶斯聚类法的Structure分析也证实了这一结果。
中图分类号:
王玲, 左示敏, 张亚芳, 陈宗祥, 潘学彪, 黄世文. 四川省稻瘟病菌群体遗传结构分析[J]. 中国水稻科学, 2015, 29(3): 327-334.
Ling WANG, Shi-min ZUO, Ya-fang ZHANG, Zong-xiang CHEN, Xue-biao PAN, Shi-wen HUANG. Genetic Structure of Rice Blast Pathogen Magnaporthe oryzae in Sichuan Province[J]. Chinese Journal OF Rice Science, 2015, 29(3): 327-334.
| 来源地 Sampling locality | 样本数量 Sample size | 纬度 Latitude (N)/° | 经度 Longitude (E)/° |
|---|---|---|---|
| 绵阳 MY | 32 | 31.47 | 104.67 |
| 营山 YS | 30 | 31.08 | 106.56 |
| 雅安 YA | 31 | 29.98 | 103.01 |
| 北川BC | 12 | 31.62 | 104.47 |
| 武胜 WS | 19 | 30.35 | 106.29 |
表1 采自四川5个地区的稻瘟病感病样品的地理位置
Table 1 Geographic location of blast-infected rice samples collected from five regions in Sichuan Province.
| 来源地 Sampling locality | 样本数量 Sample size | 纬度 Latitude (N)/° | 经度 Longitude (E)/° |
|---|---|---|---|
| 绵阳 MY | 32 | 31.47 | 104.67 |
| 营山 YS | 30 | 31.08 | 106.56 |
| 雅安 YA | 31 | 29.98 | 103.01 |
| 北川BC | 12 | 31.62 | 104.47 |
| 武胜 WS | 19 | 30.35 | 106.29 |
图1 稻瘟病菌A1和A2在荧光标记pyrms 125-126座位上的毛细管电泳峰型
Fig. 1. Capillary electrophoresis profiles of isolates A1 (up) and A2 (down) of Magnaporthe oryzae at fluorescent marker locus pyrms 125-126.
| 标记 Marker | 观测等位 基因数 Na | 有效等位 基因数Ne | Shannon’s 信息指数I | 观测杂合度 Ho | 期望杂合度 He | 哈-温平衡 偏离程度P |
|---|---|---|---|---|---|---|
| pyrms 81-82 | 7 | 1.8 | 0.840 | 0.516 | 0.439 | 0.000 |
| pyrms 99-100 | 8 | 3.2 | 1.412 | 0.943 | 0.689 | 0.000 |
| pyrms 125-126 | 7 | 2.6 | 1.225 | 0.000 | 0.615 | 0.000 |
| pyrms 233-234 | 5 | 1.6 | 0.778 | 0.072 | 0.391 | 0.000 |
| pyrms 409-410 | 12 | 7.4 | 2.154 | 0.516 | 0.869 | 0.000 |
| pyrms 533-534 | 4 | 2.1 | 0.913 | 0.274 | 0.527 | 0.000 |
| 平均值Mean | 7.2 | 3.1 | 1.220 | 0.387 | 0.588 | - |
表2 稻瘟病菌6个微卫星标记的等位基因数、杂合度及Shannon’s信息指数
Table 2 Number of alleles, heterozygosity and Shannon’s information index for six microsatellite markers of Magnaporthe oryzae isolates.
| 标记 Marker | 观测等位 基因数 Na | 有效等位 基因数Ne | Shannon’s 信息指数I | 观测杂合度 Ho | 期望杂合度 He | 哈-温平衡 偏离程度P |
|---|---|---|---|---|---|---|
| pyrms 81-82 | 7 | 1.8 | 0.840 | 0.516 | 0.439 | 0.000 |
| pyrms 99-100 | 8 | 3.2 | 1.412 | 0.943 | 0.689 | 0.000 |
| pyrms 125-126 | 7 | 2.6 | 1.225 | 0.000 | 0.615 | 0.000 |
| pyrms 233-234 | 5 | 1.6 | 0.778 | 0.072 | 0.391 | 0.000 |
| pyrms 409-410 | 12 | 7.4 | 2.154 | 0.516 | 0.869 | 0.000 |
| pyrms 533-534 | 4 | 2.1 | 0.913 | 0.274 | 0.527 | 0.000 |
| 平均值Mean | 7.2 | 3.1 | 1.220 | 0.387 | 0.588 | - |
| 来源地 Sampling locality | 观测等位基因数 Na | 有效等位基因数 Ne | Shannon’s信息指数 I | 观测杂合度 Ho | 期望杂合度 He |
|---|---|---|---|---|---|
| 绵阳MY | 6.2 | 3.1 | 1.299 | 0.406 | 0.645 |
| 营山YS | 2.3 | 1.6 | 0.445 | 0.472 | 0.281 |
| 雅安YA | 3.3 | 2.6 | 0.957 | 0.344 | 0.558 |
| 北川BC | 3.5 | 2.6 | 0.996 | 0.319 | 0.587 |
| 武胜WS | 3.2 | 2.2 | 0.780 | 0.333 | 0.439 |
| 平均值Mean | 3.7 | 2.4 | 0.895 | 0.374 | 0.502 |
表3 四川省5个稻瘟病菌群体的遗传变异分析
Table 3 Analysis of genetic variability in five populations of Magnaporthe oryzae in Sichuan Province.
| 来源地 Sampling locality | 观测等位基因数 Na | 有效等位基因数 Ne | Shannon’s信息指数 I | 观测杂合度 Ho | 期望杂合度 He |
|---|---|---|---|---|---|
| 绵阳MY | 6.2 | 3.1 | 1.299 | 0.406 | 0.645 |
| 营山YS | 2.3 | 1.6 | 0.445 | 0.472 | 0.281 |
| 雅安YA | 3.3 | 2.6 | 0.957 | 0.344 | 0.558 |
| 北川BC | 3.5 | 2.6 | 0.996 | 0.319 | 0.587 |
| 武胜WS | 3.2 | 2.2 | 0.780 | 0.333 | 0.439 |
| 平均值Mean | 3.7 | 2.4 | 0.895 | 0.374 | 0.502 |
| 来源地 Sampling locality | 绵阳(MY) | 营山(YS) | 雅安(YA) | 北川(BC) | 武胜(WS) |
|---|---|---|---|---|---|
| 绵阳MY | 0.507 | 2.496 | 4.347 | 0.940 | |
| 营山YS | 0.492 | 0.742 | 0.472 | 1.715 | |
| 雅安YA | 0.100 | 0.336 | 3.014 | 2.096 | |
| 北川BC | 0.057 | 0.528 | 0.082 | 1.317 | |
| 武胜WS | 0.265 | 0.145 | 0.119 | 0.189 |
表4 四川省5个稻瘟病菌群体间的遗传分化系数(Fst,对角线下)和基因流(Nm,对角线上)
Table 4 Pairwise population differentiation (Fst, below diagonal) and gene flow (Nm, above diagonal) among five populations of Magnaporthe oryzae in Sichuan Province.
| 来源地 Sampling locality | 绵阳(MY) | 营山(YS) | 雅安(YA) | 北川(BC) | 武胜(WS) |
|---|---|---|---|---|---|
| 绵阳MY | 0.507 | 2.496 | 4.347 | 0.940 | |
| 营山YS | 0.492 | 0.742 | 0.472 | 1.715 | |
| 雅安YA | 0.100 | 0.336 | 3.014 | 2.096 | |
| 北川BC | 0.057 | 0.528 | 0.082 | 1.317 | |
| 武胜WS | 0.265 | 0.145 | 0.119 | 0.189 |
| 变异来源 Source of variation | 自由度 df | 平方和 Sum of squares | 方差分量 Variance components | 方差比例 Percentage of variance/% | P值 Pvalue |
|---|---|---|---|---|---|
| 群体间 Among populations | 4 | 72.948 | 0.347 | 18.83 | < 0.001 |
| 群体内 Within populations | 243 | 362.806 | 1.493 | 81.17 | < 0.001 |
| 总和Total | 247 | 435.754 | 1.839 |
表5 四川省稻瘟病菌群体间和群体内的分子变异方差分析
Table 5 Analysis of molecular variance (AMOVA) within and among Magnaporthe oryzae populations in Sichuan Province.
| 变异来源 Source of variation | 自由度 df | 平方和 Sum of squares | 方差分量 Variance components | 方差比例 Percentage of variance/% | P值 Pvalue |
|---|---|---|---|---|---|
| 群体间 Among populations | 4 | 72.948 | 0.347 | 18.83 | < 0.001 |
| 群体内 Within populations | 243 | 362.806 | 1.493 | 81.17 | < 0.001 |
| 总和Total | 247 | 435.754 | 1.839 |
| 群体Population | 绵阳 MY | 营山 YS | 雅安 YA | 北川 BC | 武胜 WS |
|---|---|---|---|---|---|
| 绵阳 MY | 0.600 | 0.843 | 0.886 | 0.677 | |
| 营山YS | 0.511 | 0.772 | 0.678 | 0.924 | |
| 雅安YA | 0.171 | 0.258 | 0.874 | 0.874 | |
| 北川BC | 0.121 | 0.389 | 0.135 | 0.802 | |
| 武胜WS | 0.391 | 0.079 | 0.135 | 0.221 |
表6 四川省5个稻瘟病菌群体间的Nei’s遗传距离(GD)(左下角)和遗传一致度(GI)(右上角)
Table 6 Nei’s genetic distance (below diagonal) and genetic identity (above diagonal) among five populations of Magnaporthe oryzae in Sichuan Province.
| 群体Population | 绵阳 MY | 营山 YS | 雅安 YA | 北川 BC | 武胜 WS |
|---|---|---|---|---|---|
| 绵阳 MY | 0.600 | 0.843 | 0.886 | 0.677 | |
| 营山YS | 0.511 | 0.772 | 0.678 | 0.924 | |
| 雅安YA | 0.171 | 0.258 | 0.874 | 0.874 | |
| 北川BC | 0.121 | 0.389 | 0.135 | 0.802 | |
| 武胜WS | 0.391 | 0.079 | 0.135 | 0.221 |

图2 基于Nei’s遗传距离的四川省5个稻瘟病菌群体的UPGMA聚类分析
Fig. 2. UPGMA dendrogram of five populations of Magnaporthe oryzae in Sichuan Province based on Nei’s genetic distance.
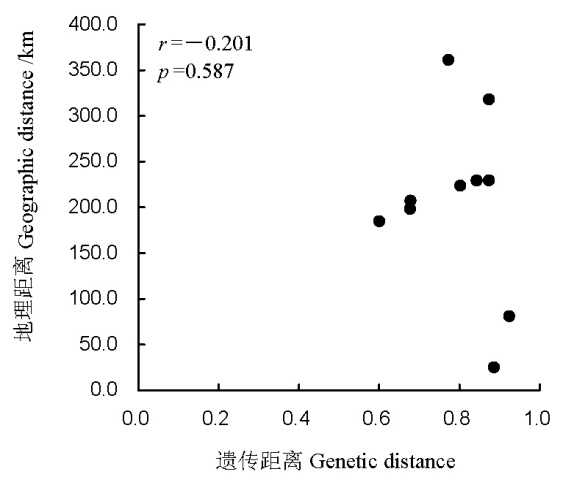
图3 四川省5个稻瘟病菌群体的遗传距离与地理距离的Mantel检验
Fig.3. Mantel test between genetic distances and geographical distances for five populations of Magnaporthe oryzae in Sichuan Province.
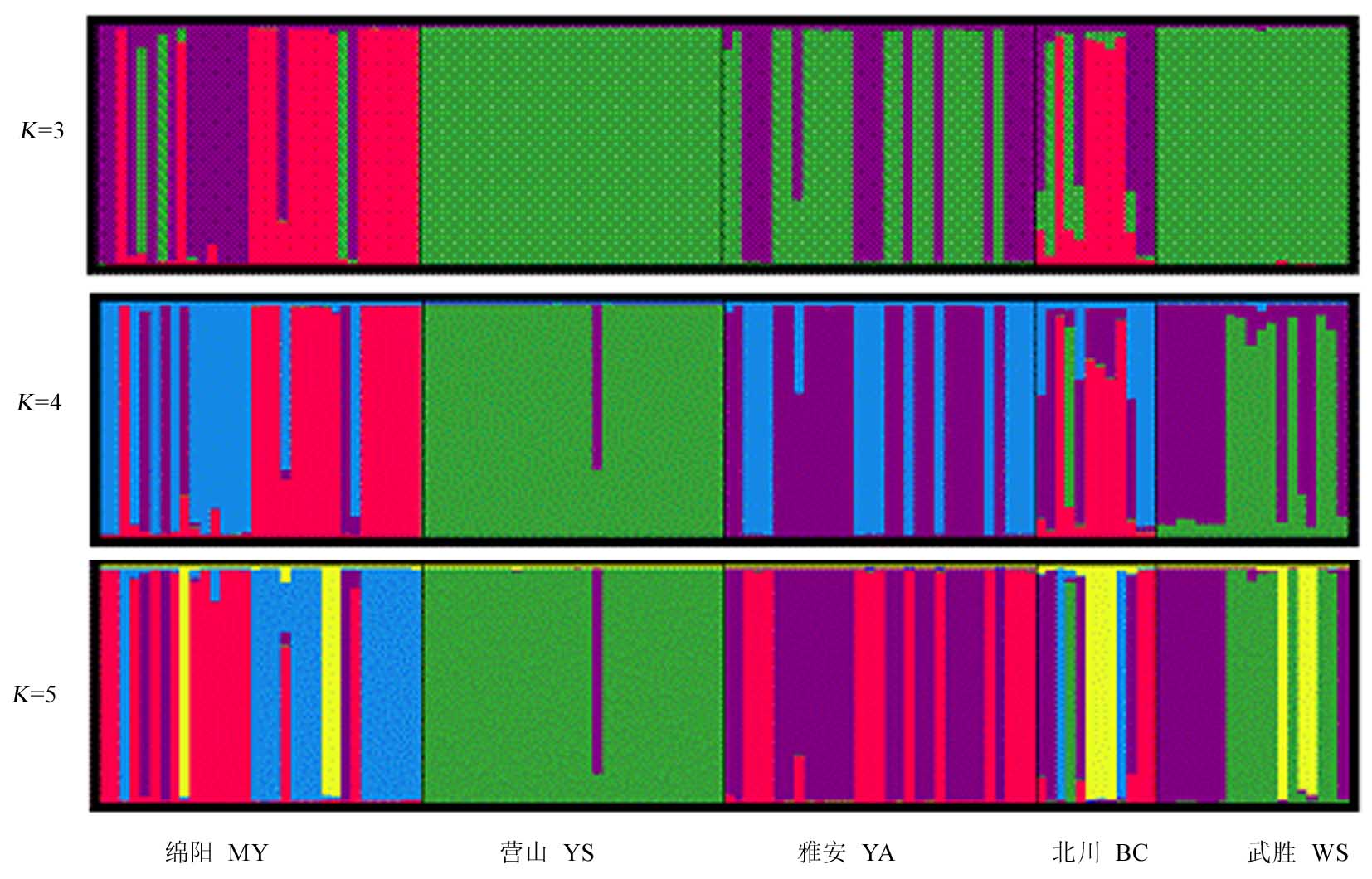
图4 基于贝叶斯聚类法的Structure分析稻瘟病菌群体遗传结构
Fig. 4. Estimated population structure of Magnaporthe oryzae population based on Bayesian clustering algorithm analysis by Structure.
| [1] | 何秀英, 刘新琼, 王丽. 稻瘟病新隐性抗病基因pi55(t)的遗传及定位. 中国科学:生命科学, 2012, 42(2): 125-134. |
| [2] | 李彬, 邓元宝, 颜学海, 等. 一个粳稻来源抗稻瘟病基因的鉴定、遗传分析和基因定位. 作物学报, 2014, 40(1): 54-62. |
| [3] | 柳武革, 王丰, 刘振荣, 等. 利用分子标记技术聚合Pi-1和Pi-2基因改良三系不育系荣丰A的稻瘟病抗性. 分子植物育种, 2012, 10(5): 575-582. |
| [4] | Xia J Q, Correll J C, Lee F N, et al.Regional population diversity of Pyricularia grisea in Arkansas and the influence of host selection.Plant Dis, 2000, 84(8): 877-884. |
| [5] | Singh Y, Kumar J.Study of genomic fingerprints profile of Magnaporthe grisea from finger millet (Eleusine coracana) by random amplified polymorphic DNA-polymerase chain reaction (RAPD-PCR).Afr J Biotechnol, 2010, 9(46): 7798-7804. |
| [6] | 吴伟怀,王玲,何艺郡,等. 稻瘟病菌群体的分子遗传学研究—广东省与江苏省稻瘟病菌群体遗传及致病型结构的比较分析. 中国农业科学, 2004, 37(11): 1628-1635. |
| [7] | Silva G B, Prabhu A S, Filippi M C C, et al. Genetic and phenotypic diversity of Magnaporthe oryzae from leaves and panicles of rice in commercial fields in the State of Goias, Brazil.Trop Plant Pathol, 2009, 34(2): 71-76. |
| [8] | Takan J P,Chipili J, Muthumeenakshi S, et al.Magnaporthe grisea populations adapted to finger millet and rice exhibit distinctive patterns of genetic diversity, sexuality and host interaction.Mol Biotechnol, 2012, 50(2): 145-158. |
| [9] | 沈瑛,Julien F, 何月秋,等. 湖南烟溪病圃稻瘟病菌的有性态及微卫星标记的遗传多样性分析. 中国水稻科学, 2004, 18(3): 262-268. |
| [10] | 李亚,刘二明,戴良英,等. 湖南稻瘟病菌群体遗传多样性与病菌致病型的关系. 中国水稻科学, 2007, 21(3): 304-308. |
| [11] | Choi J, Kim H, Lee Y H, et al.Comparative analysis of the Korean population of Magnaporthe oryzae by multilocus microsatellite typing.Plant Pathol J, 2013, 29(4): 435-439. |
| [12] | Saleh D, Milazzo J, Adreit H, et al.South-east Asia is the center of origin, diversity and dispersion of the rice blast fungus, Magnaporthe oryzae.New Phytol, 2014, 201: 1440-1456. |
| [13] | Xing J, Jia Y, Correll J C, et al.Analysis of genetic and molecular identity among field isolates of the rice blast fungus with an international differential system, Rep-PCR, and DNA sequence.Plant Dis, 2013, 97: 491-495. |
| [14] | 黄富, 叶华智, 谢戎, 等. 优质和抗稻瘟病的水稻种质资源筛选. 作物学报, 2006, 32(10): 1549-1553. |
| [15] | 王世维, 郑文静, 赵家铭, 等. 辽宁省稻瘟病菌无毒基因型鉴定及分析. 中国农业科学, 2014, 47(3): 462-472. |
| [16] | Kaye J, Milazzo J, Rozenfeld M, et al.The development of simple sequence repeat markers for Magnaporthe grisea and their integration into an established genetic linkage map.Fungal Genet Biol, 2003, 40: 207-214. |
| [17] | Adreit H, Santoso, Andriantsimialona D, et al.Microsatellite markers for population studies of the rice blast fungus,Magnaporthe grisea. Mol Ecol Notes, 2007, 7: 667-670. |
| [18] | Yeh F C, Yang R C, Boyle T.Popgene Version 1.31 Quick User Guide. Canada: University of Alberta and Centre for International Forestry Research,1999. |
| [19] | Tamura K, Stecher G, Peterson D, et al.MEGA6: Molecular Evolutionary Genetics Analysis Version 6.0.Mol Biol Evol, 2013, 30: 2725-2729. |
| [20] | Excoffier L, Laval G, Schneider S.Arlequin ver. 3. 0: An integrated software package for population genetics data analysis.Evol Bioinform Online, 2005, 1: 47-50. |
| [21] | Pritchard J K, Stephens M, Donnelly P.Inference of population structure using multilocus genotype data.Genetics, 2000, 155: 945-959. |
| [22] | Evanno G, Regnaut S, Goudet J.Detecting the number of clusters of individuals using the software structure: A simulation study.Mol Ecol, 2005, 14: 2611-2620. |
| [23] | 卢代华, 叶慧丽, 廖华明, 等. 四川籼稻区稻瘟病菌群体遗传结构. 植物保护学报, 2005, 32(1): 23-28. |
| [24] | 王艳丽, 张正光, 郑小波. 源自江苏省的稻瘟病菌营养体亲和能力与无性重组的研究. 中国农业科学, 2003, 36(6): 646-650. |
| [25] | Consolo V F, Cordo C A, Salerno G L, et al.DNA fingerprint and pathotype diversity of Pyricularia oryzae populations from Argentina.Aus Plant Pathol, 2008, 37: 357-364. |
| [26] | Park S Y, Milgroom M G, Han S S, et al.Genetic differentiation of Magnaporthe oryzae populations from scouting plots and commercial rice fields in Korea.Phytopathology, 2008, 98: 436-442. |
| [27] | Javan-Nikkhah M, McDonald B A, Banke S, et al. Genetic structure of Iranian Pyricularia grisea populations based on rep-PCR fingerprinting.Eur J Plant Pathol, 2004, 110: 909-919. |
| [28] | Hayashi N, Li C Y, Li J L, et al.In vitro production on rice plants of perithecia of Magnaporthe grisea from Yunnna, China.Mycol Res, 1997, 101: 1308-1310. |
| [29] | Rathour R, Singh B M, Sharma T R, et al.Population structure of Magnaporthe grisea from North-Western Himalayas and its implications for blast resistance breeding of rice.J Phytopathology, 2004, 152: 304-312. |
| [30] | Holsinger K E, Weir B S.Genetics in geographically structured populations: Defining, estimating and interpreting Fst.Nat Rev Genet, 2009, 10: 639-650. |
| [31] | 雷财林, 王久林, 蒋琬如, 等. 我国北方部分稻区稻瘟病菌群体遗传结构研究. 植物病理学报, 2002, 32(3): 219-226. |
| [32] | 刘振华, 李晓菲, 王园媛, 等. 云南省稻瘟病菌群体遗传结构研究. 云南农业大学学报, 2013, 28(1): 9-15. |
| [33] | 李文强, 王源超, 郑小波. 宁夏稻瘟病菌群体遗传结构研究. 南京农业大学学报, 2008, 31(4): 66-72. |
| [1] | 许丹洁, 林巧霞, 李正康, 庄小倩, 凌宇, 赖美玲, 陈晓婷, 鲁国东. OsOPR10正调控水稻对稻瘟病和白叶枯病的抗性[J]. 中国水稻科学, 2024, 38(4): 364-374. |
| [2] | 吴玥, 梁铖玮, 赵辰妃, 孙健, 马殿荣. 直播稻田杂草稻灾害发生及生态型的演变特征[J]. 中国水稻科学, 2024, 38(4): 447-455. |
| [3] | 童琪, 王春燕, 阙亚伟, 肖宇, 王政逸. 稻瘟病菌热激蛋白(HSP)40编码基因MoMHF6的鉴定及功能研究[J]. 中国水稻科学, 2023, 37(6): 563-576. |
| [4] | 陈明亮, 熊文涛, 沈雨民, 熊焕金, 罗世友, 吴小燕, 胡兰香, 肖叶青. 广谱抗稻瘟病水稻保持系赣香B的抗性遗传解析[J]. 中国水稻科学, 2023, 37(5): 470-477. |
| [5] | 李刚, 高清松, 李伟, 张雯霞, 王健, 程保山, 王迪, 高浩, 徐卫军, 陈红旗, 纪剑辉. 定向敲除SD1基因提高水稻的抗倒性和稻瘟病抗性[J]. 中国水稻科学, 2023, 37(4): 359-367. |
| [6] | 王雨, 孙全翌, 杜海波, 许志文, 吴科霆, 尹力, 冯志明, 胡珂鸣, 陈宗祥, 左示敏. 利用抗稻瘟病基因Pigm和抗纹枯病数量性状基因qSB-9TQ、qSB-11HJX改良南粳9108的抗性[J]. 中国水稻科学, 2023, 37(2): 125-132. |
| [7] | 周永林, 申小磊, 周立帅, 林巧霞, 王朝露, 陈静, 冯慧捷, 张振文, 陈晓婷, 鲁国东. OsLOX10正调控水稻对稻瘟病和白叶枯病的抗性[J]. 中国水稻科学, 2022, 36(4): 348-356. |
| [8] | 曹煜东, 肖湘谊, 叶乃忠, 丁晓雯, 易晓璇, 刘金灵, 肖应辉. 生长素调控因子OsGRF4协同调控水稻粒形和稻瘟病抗性[J]. 中国水稻科学, 2021, 35(6): 629-638. |
| [9] | 刘树芳, 董丽英, 李迅东, 周伍民, 杨勤忠. 持有Pi9基因的水稻单基因系IRBL9-W对稻瘟病菌苗期和成株期抗性差异[J]. 中国水稻科学, 2021, 35(3): 303-310. |
| [10] | 董俊杰, 曾宇翔, 季芝娟, 梁燕, 杨长登. 273份水稻种质资源的遗传多样性、群体结构与连锁不平衡 分析[J]. 中国水稻科学, 2021, 35(2): 130-140. |
| [11] | 朱华珺, 周瑚, 任佐华, 刘二明. 枯草芽孢杆菌JN005胞外抗菌物质及对水稻叶瘟防治效果[J]. 中国水稻科学, 2020, 34(5): 470-478. |
| [12] | 许赵蒙, 李利华, 高晓庆, 袁正杰, 李莘, 田旭丹, 王岚岚, 瞿绍洪. 转Pi9抗稻瘟病基因水稻株系的比较转录组分析[J]. 中国水稻科学, 2020, 34(3): 245-255. |
| [13] | 孟峰, 张亚玲, 靳学慧. 黑龙江省稻瘟病菌无毒基因AVR-Pita及其同源基因的检测与分析[J]. 中国水稻科学, 2020, 34(2): 143-149. |
| [14] | 李金璐, 张慧, 焦泽宇, 刘剑宇, 韩光煜, 卓晓轩, 罗琼. 水稻子预44和江南香糯基因组比较鉴定稻瘟病抗性相关基因[J]. 中国水稻科学, 2020, 34(1): 8-16. |
| [15] | 陈涛, 孙旭超, 张善磊, 梁文化, 周丽慧, 赵庆勇, 姚姝, 赵凌, 赵春芳, 朱镇, 张亚东, 王才林. 稻瘟病广谱抗性基因Pigm特异性分子标记的开发和应用[J]. 中国水稻科学, 2020, 34(1): 28-36. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||